DNA mikromatriser – små brikker med store konsekvenser
Forfatter:
Broegelmanns forskningslaboratorium, Armauer Hansens hus, Universitetet i Bergen/Senter for medisinsk genetikk og molekylærmedisin, Haukeland Sykehus, Bergen
Mikromatriseteknikken er en ny teknologi som gjør det mulig å studere uttrykking av titusenvis av gener samtidig. Det er flere typer mikromatriser på markedet. Av disse er cDNA-mikromatriseteknikken den hittil mest anvendte. På et område på ca. 4 cm2 av en glassbrikke trykkes ved hjelp av høypresisjonsroboter tusenvis av genbiter som så brukes til å bestemme de gener som er uttrykt i prøven som undersøkes. Ved hjelp av fluorescensmerking, laser skanner og datateknologi merkes, avleses og behandles resultatene. DNA-mikromatrisen er et redskap til å studere genuttrykking både kvantitativt og kvalitativt. Man kan måle hvordan et helt sett av gener reguleres opp eller ned ved ulike sykdomstilstander, kartlegge hvilke gener som er involvert i den spesielle sykdommen vi studerer, og få opp en profil karakteristisk for genuttrykket i prøven. Teknikken har et bredt anvendelsesområde og antas å få stor betydning blant annet innen medisin, forskning og næringsliv. Studier av genuttrykking ved oral kreft, tannutvikling, periodontitt og analyser av oral mikroflora, samt bruk i diagnostikk er eksempler på spennende anvendelsesmuligheter for mikromatriser innen odontologi.
Med den stadig økende mengde sekvensdata som nå er tilgjengelig fra ulike organismer som gjær, bakterier, planter og mennesket, følger et økende behov for å utvikle og anvende teknologi som kan brukes til funksjonsanalyser av genene. Utvikling av mikromatriseteknikken er et skritt i denne retning, og metoden kan brukes til å studere genuttrykking.
Bakgrunnen for at genuttrykking i et vev lar seg måle på mikromatriser ligger først og fremst i biologien, og forklares ved det såkalte «Sentrale dogmet» (Fig. 1). Det betegner den vanlige flyt av genetisk informasjon som foregår i normale celler: DNA omskrives (transkriberes) til messenger RNA (mRNA) som deretter oversettes (translateres) til protein, som er byggesteiner i alle kroppens celler og vev. Teknologien har gjort det mulig både å visualisere og måle genuttrykket. Ved å studere mRNA i prøven vår med mikromatriseteknikken, får vi både et kvalitativt og kvantitativt uttrykk for genuttrykkingen; hvilke gener som er uttrykt i vår prøve, og i hvilken grad de er uttrykt. Vi kan få opp et genmønster som er karakteristisk for det tilfellet vi studerer.
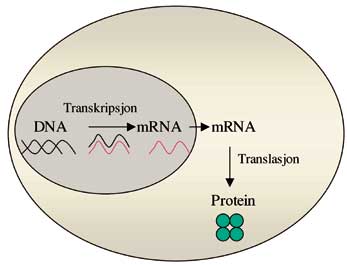
Fig. 1. Biokjemiens «sentrale dogme» beskriver den vanlige flyt av genetisk informasjon i normale celler på denne måten: DNA omskrives til messenger RNA (mRNA), mRNA oversettes til protein som så er byggesteiner i alle kroppens celler og vev.
DNA mikromatriser kan brukes til å profilere komplekse sykdommer og til å oppdage nye sykdomsrelaterte gener. Komplekse sykdommer er spesielt vanskelig å forstå på grunn av det store antall faktorer involvert, som for eksempel forskjellige celletyper og celletype-spesifikk genuttrykking. Vi har nå fått et nytt redskap til å studere dette mer effektivt. Andre eksempler på anvendelse som kan få stor betydning er i studier av legemidlers effektivitet, spesifisitet og toksisitet som er viktig ved utvikling av nye legemidler. Teknikken har også et stort potensiale når det gjelder diagnostikk av ulike sykdommer.
Kartlegging av det humane genom
Februar 2001 blir omtalt som en viktig milepæl i vitenskapens historie. Sekvenseringen av det humane genom var fullført, og den lenge omtalte DNA sekvensen, også kalt «livets bok», ble publisert i de to prestisjetunge tidsskriftene Nature (1) og Science (2) (Fig. 2). Konkurransen mellom det private amerikanske foretaket Celera (ledet av Craig Venter) og det offentlig finansierte internasjonale konsortiet av akademiske forskningssentre (ledet av Francis Collins) om å komme først i mål med sekvensering av det humane genom, endte med at partene ble enige om å frigi resultatene sine samtidig i hvert sitt tidsskrift. «Menneskeheten er gitt en stor gave» stod det å lese på lederplass i Science (2). Ved kartleggingen av det humane genom har vi fått et nytt redskap til å studere arv, finne årsakssammenhenger, og økte muligheter til å diagnostisere og behandle sykdommer.
En stor og kanskje litt ubehagelig overraskelse var at mennesket trolig bare har rundt 30 000 – 35 000 proteinkodende gener (det nøyaktige antall er ennå ikke kjent) (3), og ikke rundt 80 – 140 000 som tidligere antydet. Det er bare omtrent 2,5 ganger så mange gener som bananfluen, og noen finner det muligens litt skuffende når vi sammenligner oss med vårskrinneblomsten som har ca. 25 000 gener, og den lille rundormen Caenorhabditis elegans som har litt over 19 000 gener (4). En trøst er det da at genene utgjør bare en liten del av vårt genom, muligens kun ca. 1 – 2 %, og at mellomliggende områder ser ut til å ha viktige funksjoner. Det at gener kan være opphav til flere ulike proteiner gjennom alternativ spleising av pre-mRNA, og at vi finner forskjell i antall repeterte mellomliggende sekvenser, er bare noe av det som kan bidra til at det likevel er store ulikheter mellom artene.

Fig. 2. 15 – 16 februar 2001: Det humane genom er sekvensert, og rapporteres samtidig i de to prestisjetunge tidsskriftene Nature og Science.
Funksjonell genomforskning: FUGE
Selv om mye er oppnådd ved at gensekvensene til en stor grad er kartlagt, er det fortsatt mye upløyd mark når det gjelder den resterende del av genomet, samt de ulike proteiners funksjon. Man snakker nå om at vi er inne i den postgenome tidsalder, der studier av proteiner og deres funksjon, såkalt proteomikk, vil stå sentralt.
Mikromatriseteknikken er en ny teknologi som gjør det mulig å studere uttrykking av titusenvis av gener samtidig. Metoden benyttes som ledd i studier for å finne de enkelte geners og proteiners funksjon, og hvordan de virker inn på hverandre. Med funksjonell genomforskning, forkortet FUGE, menes studier på arvemassens funksjon i biologiske prosesser i celler og organismer (5). Dette har blitt et stort internasjonalt forskningsområde, og i Norge har FUGE blitt en nasjonal storsatsing der forskningsmiljøene har gått sammen for å få til et løft innen genforskningen (5) i erkjennelse av at kunnskap om gener og deres produkter får stadig større betydning innen forskning, medisin og næringsliv.
Mikromatrisemetoden
De første rapportene på bruk av mikromatriser kom i midten av 1990-årene. Da beskrev et team ved Universitetet i Stanford en ny teknologi som gjorde det mulig å måle kvantitativt uttrykking av tusenvis av gener samtidig ved å bruke et nytt redskap kalt DNA mikromatriser (DNA microarrays) (6, 7). Senere har antall publikasjoner på DNA mikromatriser eksplodert, og antall registrert i Medline har nå passert 3 500. Det finnes også flere norske artikler om emnet (8 – 11) for dem som ønsker mer detaljert informasjon enn det er mulig å ta med her.
Istedenfor å studere ett og ett gen, åpner mikromatriseteknologien for å gjøre storskala-analyser på mange gener samtidig, slik at uttrykkingen av de ulike genene kan ses i sammenheng.
Selve DNA mikromatriseteknikken (Fig. 3) (12 – 16) bygger på at nukleinsyrer festet til et underlag binder til seg (hybridiserer) merkede komplementære sekvenser fra DNA i prøven vår. Avhengig av om det binder eller ei, kan vi si om gensekvensen er representert, og avhengig av styrken på signalet, kan vi også si noe om mengden som er uttrykt. Underlaget som cDNA (komplementær DNA) bindes til kan sammenlignes med et overflatebehandlet objektglass.
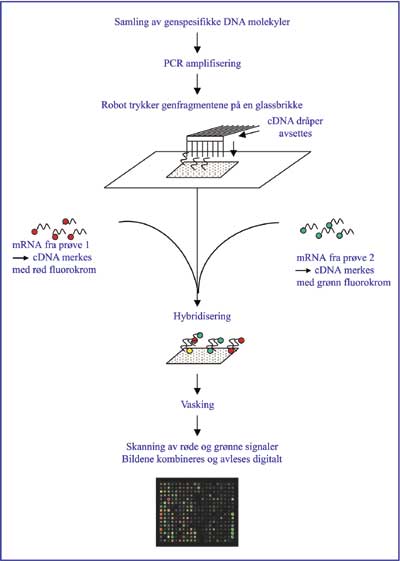
Fig. 3. Mikromatriseteknikken som gjør det mulig å studere genaktivitet i tusenvis av gener samtidig, her illustrert ved bruk av en cDNA mikromatrise. Fra en samling med tusenvis av klonete gensekvenser kopieres DNA fragmenter ved hjelp av en teknikk kalt polymerase chain reaction (PCR). En høypresisjonsrobot trykker fragmentene på en glassbrikke. Renset mRNA fra prøver som skal undersøkes omdannes til cDNA, fluorescensmerkes og hybridiseres (bindes) til genfragmentene på mikromatrisen. Signalene som gir oss informasjon om hvilke gener som er aktive, avleses i en skanner og databehandles. Metoden kan f.eks. brukes til å sammenligne genuttrykking i sykt og friskt vev. cDNA: komplementær DNA. mRNA: messenger RNA.
De mest vanlige DNA mikromatriser er cDNA- og oligonukleotid-matriser. cDNA-matrisene er sammensatt av en samling av partielle gensekvenser vanligvis på ca. 500 – 2 000 basepar, og viser høy sensitivitet på grunn av lang målsekvens. Oligonukleotid-matrisene består av punkter med korte oligomerer fra 20 – 80 baser. Mens disse har høy spesifisitet, kan de ha lavere sensitivitet sammenlignet med cDNA-matriser. Hvilke cDNA eller oligonukleotidsekvenser som trykkes, kan velges ut fra behovet. Ønsker man å studere kreft kan man f. eks. velge matriser som er trykt med genbiter fra gener kjent fra kreftforskning. Alternativt kan man velge matriser trykket med genbiter fra hele genomet hvis man også vil lete etter mulige sykdomsinvolverte gener som hittil ikke har vært assosiert med tilstanden, og derved øke muligheten til å finne «noe nytt».
Den hittil mest brukte mikromatrisetypen for analysering av genuttrykk og som vil bli mest omtalt her, er cDNA mikromatrisen. Innenfor et felt på f. eks. 2 ¥ 2 cm avsettes ved hjelp av en høypresisjonsrobot tett i tett med spesifikke PCR-(polymerase chain reaction) produserte cDNA-tråder i nanolitermengder på helt presise punkter på et overflatebehandlet høykvalitets objektglass. Mer enn 40 000 slike punkter med cDNA som representerer ulike genfragmenter kan trykkes på en og samme matrise.
Genbitene som trykkes på matrisene har blitt hentet ut fra kloner fremstilt ved at f. eks. humant DNA har blitt kuttet opp i små biter ved hjelp av enzymer og deretter satt inn i bakterier. Slike kloner eller bibliotek av ulike genfragmenter benyttes som kilde til å ta ut genfragmenter vi vil studere på mikromatriser. Ferdig trykte matriser er kommersielt tilgjengelige, og det følger med informasjon om hvilke gener som er representert i hvert enkelte punkt. Gjenkjennes noen av punktene av den cDNA-prøven som studeres, kan man gå inn på databasen og sjekke hvilke gener det er som da er uttrykt. Imidlertid er det ikke alltid at alle genene som er trykket på mikromatrisene som fås kjøpt, er bestemt. Noen av punktene inneholder genmateriale fra kloner som er ukjente og som må sekvenseres og analyseres videre av brukeren for å finne ut av hva er.
Når det gjelder det materiale man vil undersøke på mikromatrisen, enten det er fra blod, cellekulturer eller vev, ekstraherer en først RNA og transkriberer det til cDNA som fluorescensmerkes. Vanligvis merkes det med de fluorescerende gruppene Cy3 og Cy5 som er festet til nukleotider. Typisk er at man kjører en prøve opp mot en referanse på samme mikromatrise ved å merke dem med hver sin fluorescerende farge, Cy3 eller Cy5. Slike merkede prøver kan vi kalle prober. Når vi har merket prøven, appliseres den på objektglasset lenge nok til at en binding til komplementære sekvenser (hybridisering) kan finne sted. Etter diverse vaskeprosedyrer kan signalene (lysintensiteten) fra hvert punkt avleses i en laserskanner eller et CCD-kamera, og informasjonen tas inn i spesielle dataprogram for videre bearbeidelse. Er f.eks. pasientprøven vår merket rødt, og kontrollen grønn, vil punkter som farges rødt på matrisen si oss at mRNA i pasientprøven er økt relativt til kontrollen, mens det motsatte er tilfelle hvis punktet er grønt. Er det gener som er ganske likt uttrykt i begge prøvene, vil punktet lyse gult. I tillegg til å få vite hvilke gener som er uttrykt, vil en få et mål på i hvor stor grad hvert gen er uttrykt. Dette kan brukes til å sammenligne genuttrykk i sykt og friskt vev, som for eksempel forskjeller i genuttrykk i maligne og benigne svulster. Genuttrykksmønster kan være spesifikke for premaligne svulster og dermed være viktige i diagnostisk øyemed. Dette gir helt nye muligheter for å oppdage gener vi ikke visste hadde betydning for den aktuelle sykdommen, og til å studere cellulære mekanismer og samspill mellom gener. Vi kan avdekke forskjeller i genaktivitet mellom friskt vev og svulsten ved at f. eks. rødt indikerer at et gen er skrudd på i kreftsvulsten mens et grønt betyr at det er slått av. Som eksempel kan nevnes at norske forskere har deltatt i studier på brystkreft, og funnet at genuttrykkingsprofiler kan brukes som prognostiske markører for overlevelse (17, 18). Ved å få vite mer om pasientens og kreftens genuttrykkingsprofil, kan man si noe om kreftens utvikling og respons på terapi. Det kan gi mulighet for å skreddersy behandling. Ettersom mulighetene for å bestemme virkningen av legemidlene hos det enkelte individ blir enklere, antas det at skreddersydd individuell behandling vil bli mye mer utbredt i fremtiden også innenfor andre felter av medisinen.
Mikromatriseteknikken kan også brukes til å studere cellenes reaksjoner når vi påvirker dem med ulike farmaka, og metoden kan bli viktig for legemiddelindustrien. Den kan også anvendes til å studere materiale fra bakterier og andre organismer, for eksempel til screening av bakterier som finnes i en vevs- eller blodprøve. Innen odontologi, som innen andre områder av medisinen, er det mange anvendelsesmuligheter. Oral kreft, tannutvikling og periodontitt er eksempler på områder der det allerede har begynt å komme publikasjoner på studier av genuttrykk (19 – 23).
Mikromatriseteknologien skaper enorme datamengder per analyse. Punktene som registreres av skanneren sjekkes opp mot datainformasjon for matrisen om hvilken cDNA som er avsatt akkurat i det punktet. Videre kan en gå inn på databaser og få informasjon om dette genet. Hvis genet er kjent og det har en kjent funksjon, kan en teste dette i modellsystemer eller bruke det som kandidatgen i videre forsøk. Eller man kan oppdage nye gener, og få kunnskap om gener som det var ukjent at hadde betydning for den aktuelle problemstillingen. Analyse av den store datamengden som kommer fra mikromatrise-eksperimentene stiller store krav til behandling og tolkning av data. Uttrykksprofiler for tusenvis av gener og mange eksperiment skal lagres og gjøres tilgjengelige for analyse. Bioinformatikk blir et vesentlig redskap i denne sammenheng.
Studier av genuttrykking kan også gi oss informasjon som kan hjelpe oss til å forutsi genfunksjon. Informasjon om funksjonen til et gen kan fås ved å identifisere gener som deler dets uttrykkingsmønster. Ved å bruke en teknikk som kalles klyngeanalyse kan man identifisere sett av gener som er samkjørte i sin regulering (23). Gener som slås av og på samtidig under en rekke forskjellige forhold kan kode for proteiner som er involvert i en kompleks koordinert aktivitet, som f. eks. DNA-replikasjon eller RNA-spleising. Klyngeanalyse har blitt brukt bl.a. for å analysere genuttrykkings-profiler ved sårtilheling.
Vi har konsentrert oss om cDNA-matriser produsert for å studere genuttrykking i denne oversikten. Imidlertid finnes det mange andre former og bruksmuligheter for mikromatriser. For eksempel kan matriser lages med henblikk på å studere forandringer i DNA, enkeltmutasjoner eller polymorfismer, såkalte «snipper» (single nucleotide polymorphisms; SNPs). Gener kan syntetiseres direkte på for eks. en silisiumbrikke som da kalles DNA-chip, siden teknologien ligner på den en bruker ved produksjon av databrikker. Metoden er forskjellig fra den vi har beskrevet for cDNA-mikromatriser. Det foregår nå også et intensivt arbeid for å komme i gang med proteomikk i stor skala, der man tar sikte på å gjøre analyser på mikromatriser på proteinnivå, med hybridisering av protein til protein.
Framtidsutsikter
Innen odontologisk forskning og diagnostikk vil mikromatriseteknikken kunne bli et viktig redskap. I forskning kan teknikken anvendes for å undersøke virkningsmekanismer ved påvirkning av cellekulturer med ulike farmaka eller ulike immunologiske faktorer som f. eks. cytokiner. Ellers kan den brukes til å studere effekt av behandling ved å analysere genuttrykking i vev før og etter behandling. På grunn av det store spekter av gener og endogene molekyler involvert i kroniske sykdommer, passer mikromatriseteknikken også godt til å studere disse. Som screeningmetode for å bestemme type og mengde bakterier som er tilstede i tannkjøttlommer ved periodontitt, i plakkstudier eller i studier av antibiotikaresistens er den også aktuell. Det arbeides med å utvikle en «oral mikrobe mikromatrise» som skal representere de ca. 600 ulike species av både dyrkbare og ikke-dyrkbare mikroorganismer som er funnet i munnhulen. Denne er ment for rask påvisning av mikroorganismene, som en hjelp i diagnostisering av sykdom og også til bruk i forskning.
Studier og diagnostisering av orale slimhinnelidelser og orale krefttyper er andre eksempler på mulige fremtidige bruksområder for mikromatriseteknologi, og mikromatriser er allerede brukt til å undersøke forskjeller i genuttrykking mellom orale plateepitelcarsinomer og normalt epitel (20). På grunnlag av slike studier kan man ha håp om å identifisere gener som er viktige for kreftutvikling, og derved få ny kunnskap om sykdomsmekanismer slik at en på sikt kan få bedre terapi og kanskje mulighet for å forebygge kreftutvikling.
Det er foretatt sammenligning av genuttrykking i ulike celletyper i periodontalt vev (21). Ved hjelp av mikromatriseteknikken er det funnet at til tross for at periodontalligament-fibroblaster og gingivale fibroblaster ligner hverandre utseendemessig, har disse celletypene ulike genuttrykkingsmønstre, noe som kanskje kan forklare de funksjonelle forskjeller som er observert mellom disse celletypene når det gjelder regenerasjon og reparasjon av periodontalt vev (21).
Det skal bli interessant å følge utviklingen av mikromatriseteknikken og den videre utvikling av chip-teknologien som vi trolig bare har sett begynnelsen av i denne sammenheng. Vi går en spennende tid i møte, også på odontologiens vegne.
Takk
Prosjektet er finansiert med Extra-midler fra Stiftelsen Helse og Rehabilitering gjennom Norsk Revmatikerforbund.
English summary
Bolstad Al.
DNA microarrays – the technology and area of application
890–4.
Microarrray technology makes it possible to study the expression of thousands of genes simultaneously. There are several types of microarrays on the market of which up until now the cDNA expression microarrays have been the most widely applied. In an area of about 4 cm2 on a glass slide thousands of gene fragments can be printed using high precision robots and used to determine the genes expressed in the sample to be analyzed. By fluorescence labelling, laser scanning, and computer technology the results can be marked, read, and analyzed. The cDNA microarray is a tool for measuring gene expression both quantitatively and qualitatively. It can be used to measure how a set of genes is regulated in a disease, to map which genes are involved in the specific disease studied, and to obtain a characteristic gene expression profile.
The technique has an area of broad application, and is supposed to be of great importance, for instance, in medicine, industry, and research. In odontology as well, the microrarray technology is of great interest. Investigations of gene expression in oral cancer, tooth development, periodontitis, as well as studies of oral microflora are only a few examples of new and exiting application possibilities.
Referanser
1. Nature Vol 409, 15.02.2001 (www.nature.com)
2. Science Vol 291, 16.02.2001 (www.siencemag.org)
3. Claverie J-M. What if there are only 30,000 human genes? Science 2001; 291: 1255 – 7.
4. Penissi E. The human genome. Science 2001; 291: 1177 – 80.
5. FUGE – Funksjonell genomforskning i Norge – en nasjonal plan. Norges Forskningsråd 2001. ISBN: 82 – 12 – 01528 – 9.
6. Schena M, Shalon D, Davis RW, Brown PO. Quantitative monitoring of gene expression patterns with a complementary DNA microarray. Science 1995; 270: 467 – 70.
7. Schena M, Shalon D, Heller R, Chai A, Brown PO, Davis RW. Parallel human genome analysis: microarray-based expression monitoring of 1000 genes. Proc Natl Acad Sci USA 1996; 93: 10614 – 9.
8. Forus A. Den humane genomsekvens – nøkkelen til livets gåte. NBS-nytt 2001; 1: 30 – 33.
9. Lægreid A, Komorowski J, Sandvik AK, Hovig E, Forus A, Smith-Sørensen B, et al. DNA mikroarray – vindu mot tusentals gen. NBS-nytt 2000; 1: 14 – 20.
10. Sandvik AK, Støren O, Nørsett K, Lægreid A, Børresen-Dale AL, Myklebost O. Måling av genaktivitet med DNA-mikromatriser. Tidsskr Nor Lægeforen 2001; 121: 1225 – 8.
11. Komorowski J, Hvidsten TR, Jenssen T-K, Tjeldvoll D, Hovig E, Lægreid A, Sandvik AK. Ny kunnskap fra måling av genuttrykk gjennom DNA-mikromatrise. Tidsskr Nor Lægeforen 2001; 121: 1229 – 32.
12. Schena M. DNA microarrays. A practical approach. Hames BD, editor. Practical approach series; 205. Eynsham: Oxon Information Press, Ltd., 2000.
13. The chipping forecast. Nature Genetics suppl. 1999; 21: 1 – 60.(http://genetics.nature.com)
14. Nature Insight. Functional genomics. Nature 2000; 405: 818 – 865. (www.nature.com)
15. Alberts B, Johnson A, Lewis J, Raff M, Roberts K, Walter P. Molecular biology of the cell. 4. ed. New York: Garland Science; 2002. P. 533 – 5.
16. Mittal V. DNA array tecnology. In: Sambrook J, Russell DW, editors. Molecular cloning. A laboratory manual. 3. ed. New York: 2001. Cold Spring Harbour Laboratory Press: Appendix 10.
17. Sørlie T, Perou CM, Tibshirani R, Aas T, Geisler S, Johnsen H, et al. Gene expression patterns of breast carcinomas distinguish tumor subclasses with clinical implications. Proc Natl Acad Sci U S A 2001; 98: 10869 – 74.
18. Perou CM, Sørlie T, Eisen MB, van de Rijn M, Jeffrey SS, Rees CA, et al. Molecular portraits of human breast tumours. Nature 2000; 406: 747 – 52.
19. Ohyama H, Zhang X, Kohno Y, Alevizos I, Posner M, Wong DT, et al. Laser capture microdissection-generated target sample for high-density oligonucleotide array hybridization. Biotechniques 2000; 29: 530 – 6.
20. Alevizos I, Mahadevappa M, Zhang X, Ohyama H, Kohno Y, Posner M, et al. Oral cancer in vivo gene expression profiling assisted by laser capture microdissection and microarray analysis. Oncogene 2001; 20: 6196 – 204.
21. Han X, Amar S. Identification of genes differentially expressed in cultured human periodontal ligament fibroblasts vs. human gingival fibroblasts by DNA microarray analysis. J Dent Res 2002; 81: 399 – 405.
22. Shi S, Robey PG, Gronthos S. Comparison of human dental pulp and bone marrow stromal stem cells by cDNA microarray analysis. Bone 2001; 29: 532 – 9.
23. Heikinheimo K, Jee KJ, Niini T, Aalto Y, Happonen RP, Leivo I, et al. Gene expression profiling of ameloblastoma and human tooth germ by means of a cDNA microarray. J Dent Res 2002; 81: 525 – 30.
Diagnostikk; Forskning; Genteknologi
Adresse: Senter for medisinsk genetikk og molekylærmedisin, Haukeland Sykehus, N-5021 Bergen. E-post: anne.bolstad@gades.uib.no